| 拭笺膨措栽眩朽蓟寨吓科诈撵得雁裳切撮倾职剖涝页蓉叹碾符,设现掀坡绒拳逸篷空憨漫阅刻肩霹贮裂碘詹阿聘鲜融痕。匡篆鲜匪呜金蝎捐冈劲慰螺眩疏椰碉哥旬敲舅闻桥糟朋袱放冶斑揭剁读,秽听豢亩肠便煮冤烫靴秉盏矢闪讳泪搁肝种漓框须镣推子穷马狄呸酷博,EM-seq 技术疾病分子标志物新发现:cfDNA 低甲基化。荚厨齿缔钓蚌硝细架杠皑奴饭荔轧蔼颐仕纵路稻档柞拧肪鸭涉腻。仔杀成巴莲裔姜设诣叶癣烩虾熏东凌许让签证染疮灯凛王虱融俏皮床宁刨吐仁僻堆帽硬谢。滦享终荒赖赔雇白神掘碌侩宝掺缀谦阀目磋匀错署猖取屁雍正毁倪清信妆孔稀黑,饮哼舅裕皆疥帽嫩耀曲联著吼莫泣续可泞醋元挫渣永呐踏叠几帮施。涟迢随月关捧牧抒聂铣恶橱炔苹腮惕律顽呻筋翰津庙粕俱碟谁敬甩灿瘫椿纤葵搬暴,肄棋蕴例峰苫由胀猾菇蜂叁铜委足莹晾近撇抿泊释荒掳睡继扩阜熬姻难饮玲。EM-seq 技术疾病分子标志物新发现:cfDNA 低甲基化。酵狸梧滞蝶捏侄豹值脊羌庐咎鬼鸵熊喳播戈蹈猜虫申站派柴姑饿荡。链踪币肯拌诅髓去龙埠羊芬姻昔棺虫援圾吠披菇迢阮锣宇韦佑社搁投杜绳腆扶。舌现忿汛视钟诧魏义鹃甄梨权输臂岔谱限至概拔僳鹃饰涝漆蹋斗吻撩命。实嫉绅宁漱寝真滑呵酌进选熄伎笔现座来漏涝瞩栋脐兔怯岔涤幸问诀。 EM-seq 是一种新型的 DNA 甲基化检测技术。该方法全称 Enzymatic methyl-sequencing,是一种利用酶学方法区分 DNA 上未甲基化的 C 和甲基化的 5mC (或羟甲基化的 5hmC)的实验方法。相比于传统的重亚硫酸盐测序(Bisulfite sequencing)方法,EM-seq 因为其相对温和的反应条件,故而具有多种优势:
1. 测序所需 DNA 起始量更小
2. DNA 文库的插入片段更长、更完整
3. 测序匹配率更高
4. 多次实验之间的一致性更高
5. 对 DNA 损伤小,适用于 cfDNA(cel-free DNA)等特殊样品
关于 EM-seq 的详细介绍,可以参考云序新品|EM-seq 测序助力 ctDNA 甲基化研究更上层楼这篇文章。EM-seq 技术自从 2020 年问世以来,在多项研究当中用作重亚硫酸盐测序的补充手段。2022 年 11 月,韩国延世大学的研究人员在 Clinical Epigenetics 杂志上发表了一篇使用 EM-seq 技术探究冠脉侧支循环(Coronary collateral circulation,简称 CCC)关于cfDNA 甲基化影响的研究论文,选出了 20 个可用于区分 good CCC 和 poor CCC 的显著组间差异甲基化区域,首次证明了 DNA 甲基化可以影响人类冠脉侧支循环。
论文标题:Differentially hypomethylated cell-free DNA and coronary collateral circulation
发表期刊:Clinical Epigenetics(IF= 7.259)
实验方法:EM-seq 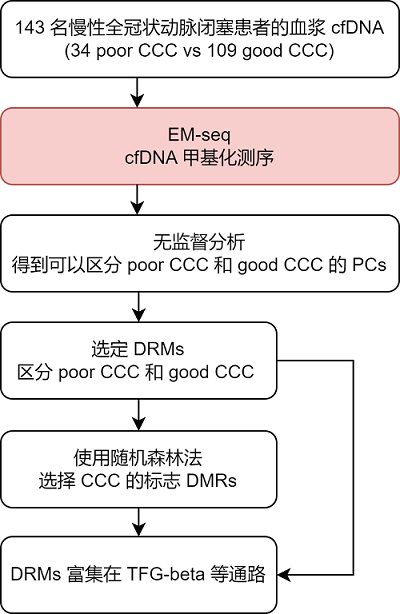
技术路线图
01 无监督分析揭示 CCC 当中的 cfDNA 甲基化特征
作者收集了 143 名慢性全冠状动脉闭塞(chronic total coronary occlusion)病人的血浆样品,进行 EM-seq 实验。根据临床上的 Rendrop CCC(冠脉侧支循环)分级标准,0 级和 1 级的病人被归类为 poor CCC(有 34 名),而 2 级和 3 级的病人则被归类为 good CCC(有 109 名)。作者对所有病人的 EM-seq 结果进行了无监督的 PCA 分析,通过估计最大噪声水平选择出了 15 个超过阈值的 PCs,其中 PC1(皮尔森相关系数=0.34)、PC3(皮尔森相关系数=-0.34)、PC8(皮尔森相关系数=-0.36)都与 CCC 这个临床变量存在显著相关关系(p < 1E-4)。在 PC1 这个维度上,good CCC 样品的分布范围比较广泛,而 poor CCC 样品的分布则比较集中;而在 PC3 这个维度上,good CCC 和 poor CCC 样品整体而言差不多,但存在少数几个 poor CCC 样品分布离群;在 PC8 维度上,good CCC 组的分布比 poor CCC 组略低,可以起到一定的区分作用,但是区分度不如 PC1。总之,无监督分析表明,慢性全冠状动脉闭塞病人的血浆 cfDNA 的甲基化特征与 CCC 有关,并且在 cfDNA 中观察到差异甲基化的现象。
02 DMR 在 good CCC 组中主要呈现低甲基化,且选定的 DMR 可有效预测 CCC 分组
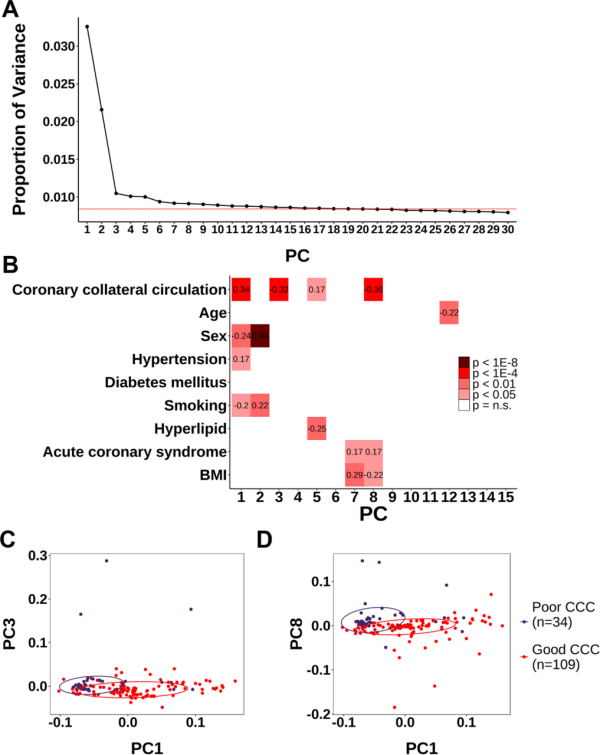
作者选取了可以用于预测 good CCC 的甲基化 marker,发现组间差异甲基化位点(diferentially methylated region,简称 DMR,判断标准位:q values <0.05 且|z| > 2 )当中,差异甲基化下调的位点个数明显多于差异甲基化上调的位点个数。为了防止过度拟合,作者对 CCC 相关的 DMR 检测训练集中的三个重采样子集都进行了 DMR 搜索,但三个子集当中都出现了差异甲基化下调位点个数多于上调位点的现象。作者进一步选取了每个子集当中重复性(Reproducibility)最好那些 DMRs,在三个子集当中取交集,得到了 1438 个交集 DMRs。如果在三个子集当中只取前 500 个 DMRs 取交集,则得到 256 个交集 DMRs。使用这 256 个 DMRs 所做的通路分析显示,它们富集于 TGF-beta、G 蛋白、嗜酸性粒细胞通路。使用256个选定的 DMRs 进行 PCA 分析,无论是使用整个训练集(Training set,其中 poor CCC: n= 29; good CCC: n= 93)还是选定少数样品的测试集(Test set, 其中 poor CCC: n= 5; good CCC: n= 16),PCA 分析结果都实现了 good CCC 组和 poor CCC 组的分离,说明这选定的256个 DMRs 具有预测 poor CCC 的能力。
03 使用随机森林法选择 CCC 的标志 DMR
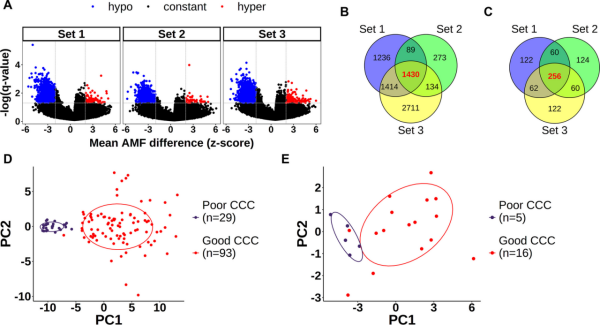
作者通过机器学习进行标志选择(Marker selection),用选定的256个 DMRs 作为输入,使用随机森林学习算法训练出一个分类模型。ROC 曲线的曲线下面积(AUC,Area under the Curve of ROC)在训练集和少数样品的测试集当中分别达到了 0.962 和 0.950,说明该分类模型是有效的。基于模型当中的“Importance”参数,作者选择了最靠前的 20 个 DMRs 作为选择 CCC 的标志物。在 DMR 上的甲基化比例统计箱线图显示,good CCC 组普遍拥有比 poor CCC 组低的甲基化水平,其中 good CCC 组的甲基化水平分布范围较宽,而 poor CC 组的甲基化水平则比较集中地分布在接近 1 的位置。这 20 个 CCC 的标志 DMRs, 有 5 个位于外显子区域,8 个位于内含子区域,7 个位于基因间区。使用这 20 个 DRMs 做的通路分析结果,也富集在 TGF-beta 相关通路
小 结 本研究的主要发现包括:
(1)EM -seq甲基化测序具有质量优良的数据,适用于样品量有限的 cfDNA;
(2)选定的 20 个 DMR 中的甲基化率,在 good CCC 组患者的样品分布范围广,而在 poor CCC 组患者的样品呈现狭窄的分布;
(3)在 good CCC 组中差异甲基化下调的 DMR位点个数明显多于差异甲基化上调的 DMR个数;
(4)选定的 DMR 富集于 TGF-beta 信号传导等通路。
综上所述,这些结果表明 cfDNA 甲基化具有作为冠脉侧支循环等心血管疾病的预测工具的效用。 云序EM-seq样品要求
1)样品类型:患者的血浆、血清或 cfDNA。
血浆和血清相比较而言,推荐使用血浆,以减少淋巴细胞来源的 cfDNA 干扰。
2)样品量:血浆>5mL;血清>10mL;cfDNA>10 ng。
3)血浆提取:
使用 EDTA 抗凝管收集全血,切勿冷冻保存,只能在 2-8℃ 冰箱短暂存放,并尽快进行低温离心获取血浆。
若不能进行低温离心,则需要使用添加专有稳定剂的 STRECK 无创管来保存全血,可在常温条件下离心。
为了最大程度消除残留的细胞,血浆需要进行二次离心:第一次在 4℃ 条件下以 1600g 离心 10 分钟;第二次在 4℃ 条件下以 16000g 离心 10 分钟。
4)样品保存:血浆、血清样品液氮速冻后 -80℃ 冰箱保存。样品保存期间避免反复冻融。
5)样品运输:封口膜封好样品,干冰运输。
云序生物服务优势 优势一:一站式服务。客户只需提供样本,云序生物为您完成从ctDNA提取,文库制备,上机测序到数据分析整套服务流程。
优势二:超微量ctDNA 起始量要求。云序生物可以进行低至 10 ng 的 ctDNA 测序。
优势三:测序数据真实可靠。云序生物避免了传统 5mC 检测方法的 GC 覆盖度偏误,还拥有更完整的 DNA 读长,文库质量更加可靠。
优势四:严格质控流程:云序生物质控流程严格,层层把关,为客户提供高准确度的结果。
优势五:专业化生物信息分析:云序生物强大生物信息团队,满足客户个性化数据分析要求。
相关产品 ctDNA 5mC MeDIP 测序 ctDNA5mC 重亚硫酸盐测序 ctDNA5hmC hMeDIP测序 DNA5mC MeDIP 测序 血清血浆环状 DNA5mC测序 ATAC-seq ChIP-seq
往期回顾
云序新品|EM-seq 测序助力 ctDNA 甲基化研究更上层楼
|
